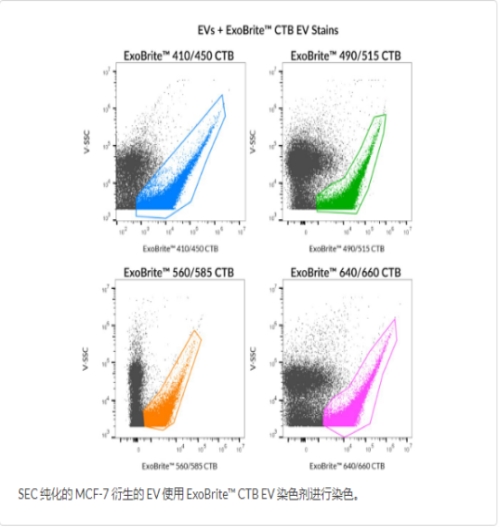
与ExoBrite™膜联蛋白EV染色剂类似,这些基于WGA的缀合物与EV具有广泛的兼容性!对来自所有9个测试细胞系的EV进行了染色验证!此外,ExoBrite™WGAEV染料比疏水膜染料更不易聚集,可用于对纯化的和珠结合的EV进行染色.ExoBrite™WGAEV染色剂的优点:·优化配制的WGA缀合物,用于对纯化或珠结合的EV进行染色·广泛的兼容性,从所有9个测试源中分离出染色的EV·用于流式细胞术的明亮信号和低背景染色·与抗体共染色兼容·有4种颜色可供选择,可实现灵活的实验设计ExoBrite™410/450WGAEV染色套件ExoBrite™490/515WGAEV染色套件ExoBrite™560/585WGAEV染色套件ExoBrite™640/660WGAEV染色套件使用ExoBrite™STORMCTBEV染色套件超越衍射极限由于外泌体和EV的尺寸小以及光学显微镜的分辨率限制,通过成像表征它们仍然是一个挑战。
·使用qPCR选择性检测活细胞·比PMA更好地区分细菌的活/死·死细胞特异性染料,与DNA结合·光活化后与DNA共价结合·20mM水溶液,100uL体积·λAbs=464nm(光解前);λAbs/λEm=~510/~610nm(含DNA/RNA,光解后)热门产品GelRed核酸凝胶染色剂——超灵敏且环保的EtBrDNA/RNA凝胶染色替代品!产品描述GelRed是一种超灵敏、极其稳定且对环境安全的荧光核酸染料,旨在替代剧毒的溴化乙锭(EtBr),用于琼脂糖凝胶或聚丙烯酰胺凝胶中的dsDNA、ssDNA或RNA染色!
·比EtBr更安全:无致突变性且处置无危险·比EtBr和SYBRSafe更灵敏·室温下稳定,可微波加热·简单的预制或电泳后凝胶染色,无需脱色·取代EtBr,无需更改设备或光学设置·与下游凝胶纯化、限制性消化、测序和克隆兼容GelRed:EtBr的替代品GelRed比EtBr灵敏得多,可用于通过预制或凝胶后染色对琼脂糖凝胶中的dsDNA、ssDNA或RNA进行染色,而无需脱色!GelRed还可用于通过凝胶后染色对聚丙烯酰胺凝胶进行染色。
上海牧荣生物科技有限公司是一家着力于研究进出口代理的公司, 经过多年的坚持不懈与努力,公司在业内也算是有属于自己的一片天。 公司多年来一直坚持为客户提供专业、快捷、周到的服务,愿与业内同仁共同致力于行业的进步。 公司主营产品有:biotium,我们在这里等待您的到来!
biotium
直接随机光学重建显微镜(dSTORM)等超分辨率显微镜技术突破了传统光学显微镜的衍射极限,允许电动汽车等亚细胞结构的单分子分辨率!ExoBrite™STORMCTBEV染色剂专为EV的STORM成像而开发,并提供四种经过STORM验证的CF染料!ExoBrite™560/585已在ONINanoimagerSMarkII上针对dSTORM进行了验证,可用于研究精细形态细节以及与EV生物标志物(如四跨膜蛋白CDCD63和CD81)进行共染色。
我司主营进出口代理领域的企业,主要以biotium为主要产品,公司位于上海市嘉定区安亭镇新源路155弄16号新源商务楼718室,更多产品信息详情请上http://www.murongbio.cn/查看。上海牧荣生物科技有限公司愿与社会各界朋友共同合作、共创双赢、共创精彩明天!
我们继续为不想改变实验方案的既定用户提供DMSO中的GelRed。我们还提供GelRed琼脂糖和GelRedPrestainPlus6X上样染料!应用:·DNA或RNA电泳·预制或电泳后染色·琼脂糖、聚丙烯酰胺、DGGE、EMSA、PFGE或甲醛凝胶·限制性内切酶图谱·PCR扩增子检测·Southern/Northern印迹·DNA带纯化、克隆和测序·彗星试验·毛细管电泳·氯化铯梯度Mix-n-Stain™CF染料抗体标记试剂盒——使用我们的一种CF染料在短短15分钟内标记您的IgG抗体,无需纯化步骤!